El antibiograma puede interpretarse
por medio de valores cualitativos como sensible, intermedio o resistente basado
en la Concentración Mínima Inhibitoria (MIC) de antimicrobiano que inhibe el
crecimiento bacteriano. Sin embargo, en la actualidad se prefiere realizar una
lectura interpretativa basada en el conocimiento de los diferentes mecanismos
de resistencia, permitiendo predecir el riesgo de resistencia a otros
antibióticos que in-vitro aparecen como sensibles.
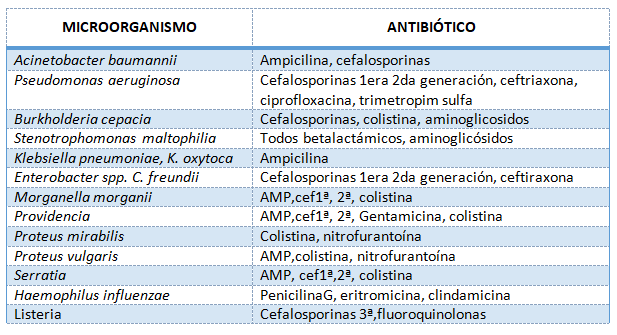
RESISTENCIA NATURAL DE ESPECIES DE ANTIBIÓTICOS
INTERPRETACIÓN DEL ANTIBIOGRAMA EN BACILOS GRAM NEGATIVOS
Betalactámicos:
- Alteración del sitio blanco de acción: producción de proteínas de unión a penicilinas (PBPs) adicionales de baja afinidad por el antibiótico ó modificaciones estructural en las PBPs originales generando una enzima con muy baja afinidad por el antibiótico.
- Trastornos de la permeabilidad: El mecanismo más importante es el que se da por mutantes deficientes en una o más porinas de la membrana externa. Como ejemplo de ello es la resistencia al imipenem en cepas de P. aeruginosa donde la deficiencia de una porina por donde ingresa el antibiótico genera resistencia a algunos carbapenémicos.
- Eflujo activo e hidrólisis enzimática mediada por betalactamasas: es el mecanismo de resistencia más reconocido de los bacilos Gram negativos a los betalactámicos, generando la hidrólisis enzimática de penicilinas, cefalosporinas y en ocasiones a carbapenémicos.
Aminoglicósidos:
Si bien las
betalactamasas son los mecanismos enzimáticos más frecuentes, existen otras
enzimas que generan resistencia concomitante a otras familias de antibióticos.
Algunas metilasas, acetiltransferasas, nucleotidiltransferasas y
fosfotransferasas que inactivan en forma efectiva los aminoglicósidos.
Quinolonas:
La
resistencia a quinolonas en enterobacterias se alcanza por acumulación de
mutaciones en los genes de la topoisomerasa, especialmente gyrA y parC. Estas
mutaciones se concentran en una región llamada QRDR. Otro mecanismos de
resistencia es la hiperexpresión de bombas de expulsión o algunas alteraciones
en porinas causando algunos niveles de resistencia bajo. Además, se han
descrito 3 mecanismos de resistencia residente en plásmidos: proteínas de
resistencia a quinolonas (Qnr) que protegen a la DNA girasa; la enzima
modificante de aminoglucósidos Aac (6′)-Ib-cr que le confiere resistencia
cruza a las quinolonas y adicionalmente un sistema de eflujo.
Trimetropim-Sulfametoxaxol:
Estos
compuestos inhiben competitivamente la incorporación del ácido
para-aminobenzoico en el ácido tetrahidropteroico, un precursor del ácido
fólico, mediante la interferencia de la enzima dihidropteroato sintetasa. La
resistencia a sulfonamidas es producida ya sea por un aumento de la síntesis
del ácido para-aminobenzoico, mutaciones en la dihidropteroato sintetasa o por
un mecanismo enzimático codificado en plásmidos que hacen inefectiva la acción
de las sulfonamidas.
Pseudomonas aeruginosa:
P. aeruginosa tiene
una resistencia natural a múltiples antibióticos y fácilmente adopta
resistencia a otros a través de la adquisición de elementos genéticos móviles o
mutaciones. La resistencia que presenta este microorganismo se debe, a
mecanismos como la producción abundante de enzimas inactivadoras, incluyendo la
expresión natural de AmpC cromosómica, la impermeabilidad de la membrana, y la
presencia intrínseca de bombas eflujo, principalmente Mex-AB-OprM (Tabla 4).
Todos estos mecanismos favorecen el fácil desarrollo de multidrogorresistencia
en este grupo de microorganismos.
El fenotipo silvestre de P.
aeruginosa es susceptible a quinolonas, aminoglicósidos, ureidopenicilinas,
ceftazidima y cefalosporinas de cuarta generación, además, sensibilidad a
aztreonam y carbapenémicos (excepto ertapenem).
INTERPRETACIÓN DEL ANTIBIOGRAMA EN COCOS GRAM POSITIVOS
A diferencia
de los bacilos Gram negativos, la resistencia en este grupo de microorganismos
está asociada a cambios estructurales en la pared celular o en componentes
citosólicos como los ribosomas y no a mecanismos enzimáticos. Los ejemplos más
representativos son aquellos mecanismos de resistencia expresados por: Staphylococcus
spp. , Enterococcus spp. y Streptococcus spp.


No hay comentarios:
Publicar un comentario